本文参考:

导读
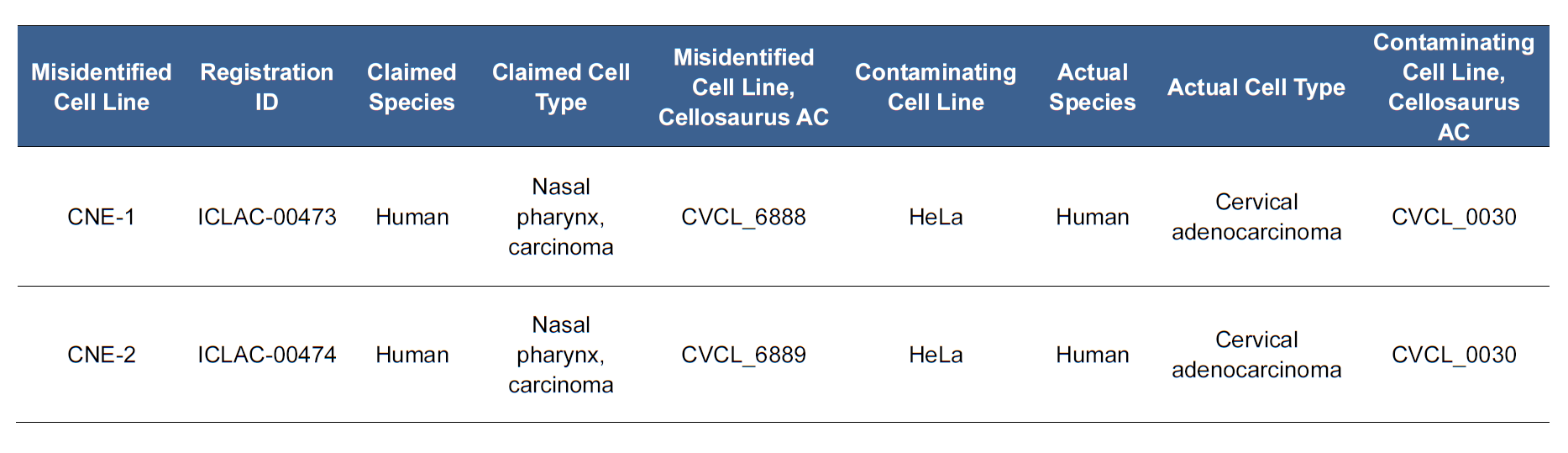
●CNE1、CNE2细胞株的建立,为我国鼻咽癌病因学研究、免疫学诊断及临床防治提供了良好的细胞实验模型。两者来源于不同个体:CNE1来源于一名女性高分化鳞癌患者;CNE2来源于一名男性低分化鳞癌患者。
(图片来源:ICLAC Database)
●但自2008年起,陆续有研究者发现这两株细胞株的STR分型结果呈现高度一致性,而且与HELA的STR数据具有较高相似度,此后又有多篇文献证实此发现。CNE1、CNE2被HELA污染,但并非完全来源于HELA,实为HELA与其他未知来源细胞的混合物。
1
CNE1细胞株由中国医学科学院、中山医学院协作建立。1975年8月,细胞株从一名58岁女性鼻咽癌患者的鼻咽部肿物的活检组织建立。病人术前诊断为鼻咽癌,病理诊断为高分化鳞癌。组织块培养10天后,少数组织周边开始出现上皮样细胞。在第12代后,上皮样细胞中开始出现梭形细胞。此后选择全为上皮样细胞传代,称CNE(即CNE1)细胞株;全为梭形细胞传代,则称CNF细胞株。CNE1未检出EB病毒的核抗原(EBNA)。
CNE2细胞株由中国医学科学院、湛江医学院协作建立。1980年,细胞株从一名63岁男性鼻咽癌患者的活检组织建立。病理诊断为低分化鳞癌。接种一周后,上皮样细胞从组织块周边长出。接种两周后,开始出现纤维细胞,且生长占据优势,交替使用刮脱和胰酶的方法逐步去除纤维细胞。第四代开始细胞全部为上皮样细胞,且呈单层生长。第三代细胞EBNA检测呈阳性,但此后的传代细胞EBNA检测为阴性。
2
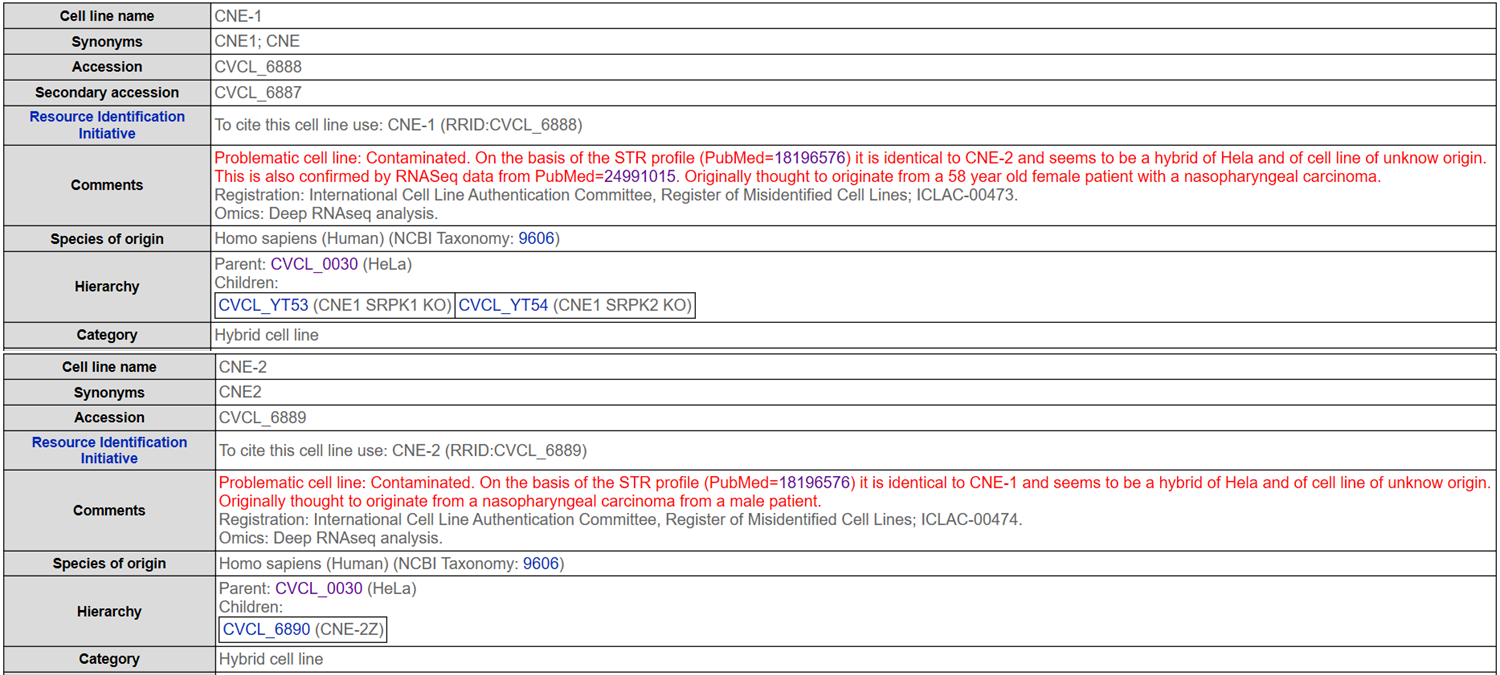
2008年,SylviaYat-Yee Chan等人的文章(PMID为18196576)报道,CNE1与CNE2的STR分型结果高度一致。更重要的发现是,两者的分型数据与HELA具有相似性,他们将两株细胞株及HELA的分型数据进行对比分析,发现在检测的15个位点中,CNE1、CNE2在每个位点的数据都至少包含一个HELA的等位基因,即有些位点(vWA、D8S1179、D7S820、CSF190、D19S433)并非与HELA基因型相同,仅含有HELA的一个等位基因。基于此发现,他们综合多种方法分析推测,CNE1、CNE2为HELA与其他未知来源细胞的混合物。
(图片来源:
2014年,Michael J. Strong等人应用High-ThroughputRNA Sequencing Analysis对包括CNE1、CNE2在内的多种鼻咽癌细胞株进行SNVs分析时发现,CNE1虽然被HELA污染,但并非完全来源于HELA。这也证实了SylviaYat-Yee Chan等人的发现。
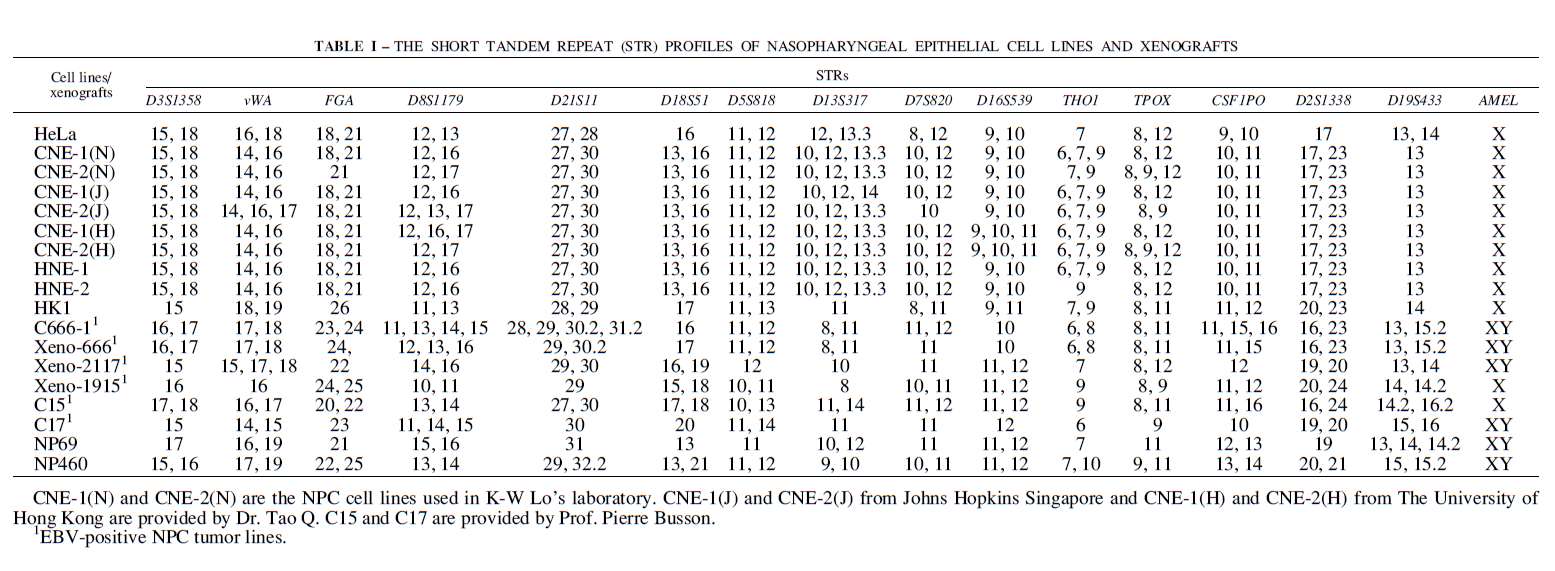
(图片来源:Sylvia Yat-Yee Chan et al., 2008)
2015年,FangYe等人同样对CNE1、CNE2进行STR分型,实验结果与SylviaYat-Yee Chan的结论一致。2017年,Yaqing Huang、Xiaocui Bian的文章也证实这两株细胞株被HELA污染。
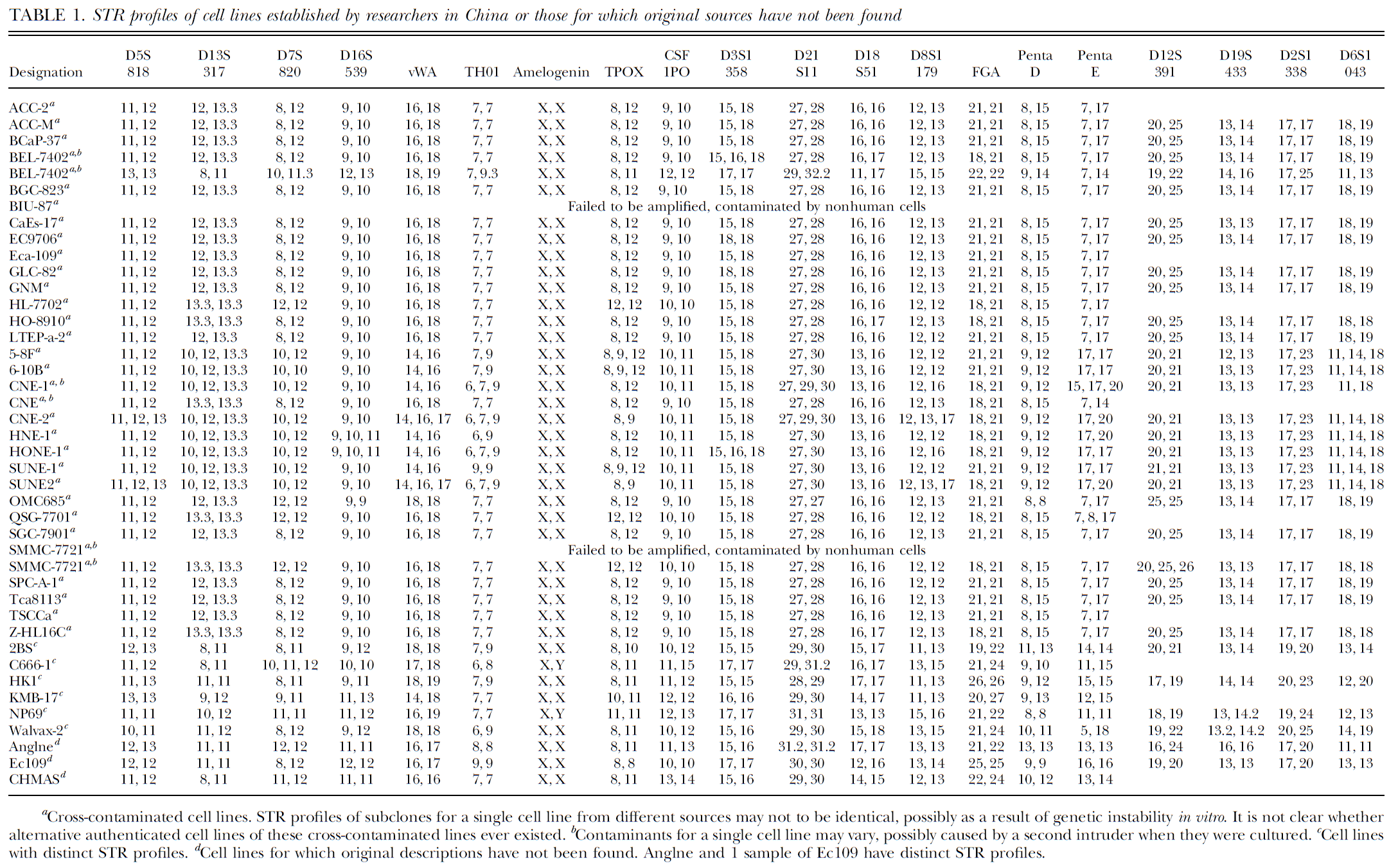
(图片来源:
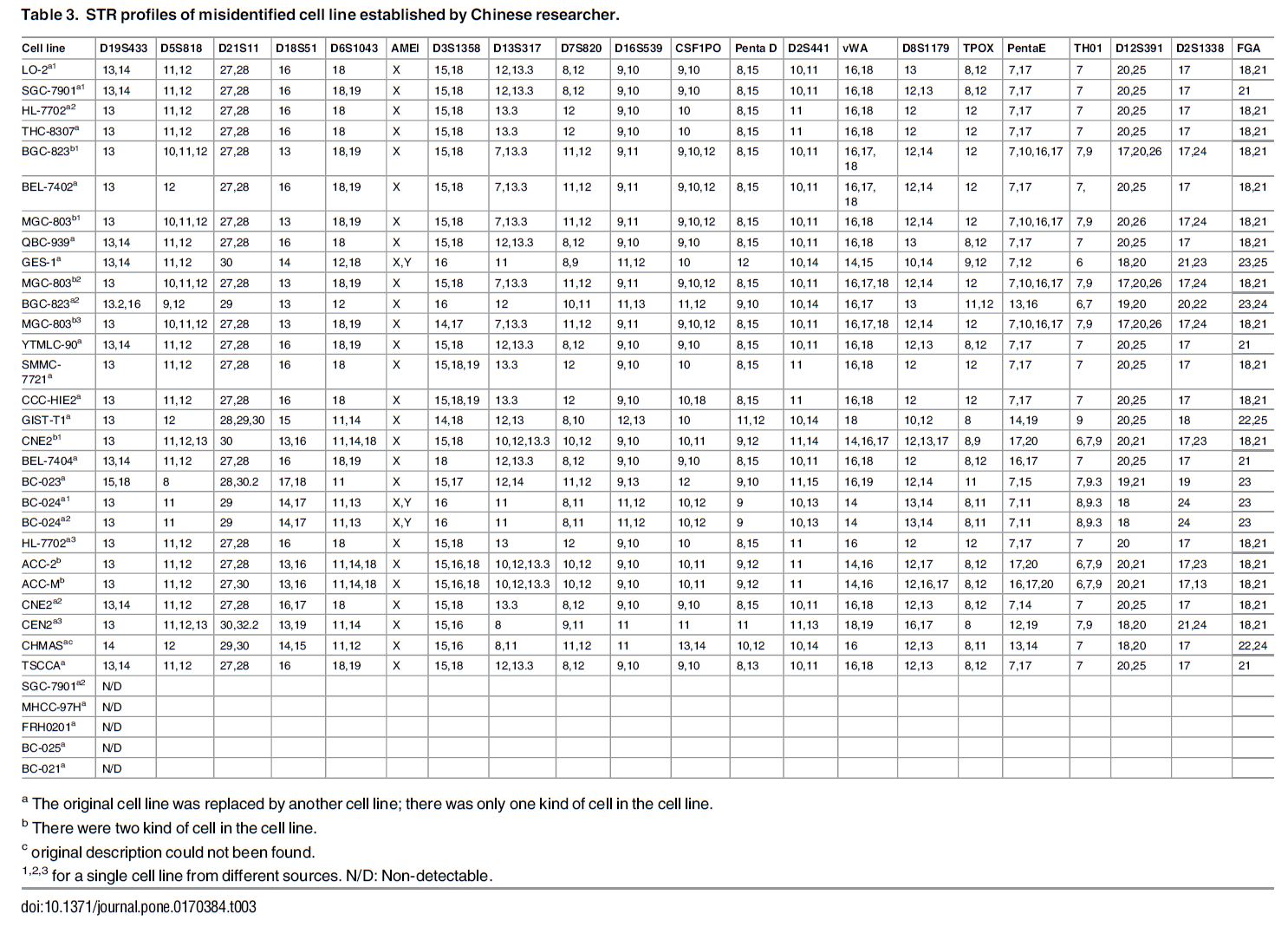
(图片来源:
3
截止目前,鉴达受理CNE1、CNE2细胞株鉴定各有5株。与ExPASy、DSMZ数据库重新比对确认,CNE1实际鉴定结果:4株与文献报道一致,为HELA与未知来源细胞的混合物,1株为非人源细胞;CNE2实际鉴定结果:3株为HELA与未知来源细胞株的混合物品,1株为HELA,1株为无匹配结果。鉴达实际检测结果显示,与ExPASy、ICLAC数据库一致,提示CNE1、CNE2被HELA污染。
参考文献:
1.中国医学科学院,人体鼻咽癌上皮样细胞株和梭形细胞株的建立,肿瘤防治研究,3:18~24,1978。
2.中国医学科学院,从低分化鼻咽癌病人建立鼻咽癌上皮细胞株,癌症,2(2):70~72,1983。
3.SylviaYat-Yee Chan et al., Authentication of nasopharyngeal carcinoma tumor lines.Int. J. Cancer. 2008 May1;122(9):2169-71.
4.Michael J Strong et al., Comprehensive High-Throughput RNA Sequencing Analysis RevealsContamination of Multiple Nasopharyngeal Carcinoma Cell Lines With HeLa CellGenomes. J Virol. 2014Sep;88(18):10696-704.
5.FangYe et al., Genetic profiling reveals an alarming rate of cross-contamination among human cell lines used in China. FASEBJ. 2015 Oct;29(10):4268-72.
6.YaqingHuang et al., Investigation of Cross-Contamination and Misidentification of 278Widely Used Tumor Cell Lines. PLoS One.2017 Jan 20;12(1):e0170384.
7. Bian X et al., A Combination of Species Identification and STR ProfilingIdentifies Cross-contaminated Cells from 482 Human Tumor Cell Lines. Sci Rep. 2017 Aug 29;7(1):9774.
苏州鉴达采用STR分型技术,针对人源细胞20个位点进行检测,3500型、3130XL型遗传分析仪进行毛细管电泳,与DSMZ、Cellosaurus、ATCC等数据库进行比对,出具权威英文报告,为全国近200家科研机构提供高质量服务,多家单位已利用鉴达细胞株鉴定服务在高水平期刊(如Clinical Cancer Research、Nature Commnications、Int J Cancer等)发表论文。
如需了解更多鉴达生物相关细胞株鉴定信息,可扫码关注官方公众号。
微信:
Selected publications with Impact Factor >5.0 using our cell line authentication service
[1]. Ren A et al., Int J Cancer. 2019 Jul1;145(1):154-163.
[2]. Shen Y et al., Int J Cancer. 2019 Feb 1;144(3):651-664.
[3]. Xu H et al., Clin Cancer Res. 2019 Mar15;25(6):1923-1935.
[4]. Li B et al., Stem Cells. 2019 Feb 37(2):247-256.
[5]. Shi J et al., Mol Pharm. 2018 Sep4;15(9):4139-4147.
[6]. Li B et al., Oncogene. 2018 Oct 37(41):5508-5519.
[7]. Yuan Y et al., Int J Cancer. 2019 Apr1;144(7):1584-1595.
[8]. Yu J et al., J Hepatol. 2018 Jun 68(6):1214-1227.
[9]. Zhang N et al., Cell Death Differ. 2019 May 26(5):843-859.
[10]. Du L et al., Cell Death Dis. 2018 Jun18;9(7):714.
[11]. Zhou P et al., J Exp Clin Cancer Res. 2017Sep 2;36(1):115.
[12]. Wang X et al., Int J Cancer. 2017 Oct1;141(7):1422-1433.
[13]. Li Y et al., Nat Commun. 2017 Aug24;8(1):347.
[14]. Wang C et al., Carcinogenesis. 2017 Feb1;38(2):168-176.
[15]. Wang X et al., Cell Death Dis. 2016 Dec8;7(12):e2517.
[16]. Li P et al., Oncogenesis. 2017 Apr24;6(4):e322.
[17]. Du J et al., Mol Cancer Ther. 2018 Feb 17(2):419-431.
[18]. Chen L et al., Nat Commun. 2017 Jul17;8:16066.
[19]. Li H et al., Cancer Lett. 2018 Sep 28;432:121-131.