本文参考:ExPASy Database、ATCC Database、NSTI Database

导读
● FHC(Fetal Human Colon)细胞株为人正常结肠上皮细胞。它的建立对结肠相关的基础实验具有重要意义,尤其在揭示结肠癌病因学研究中,为实验人员提供了一个合适的体外实验模型。
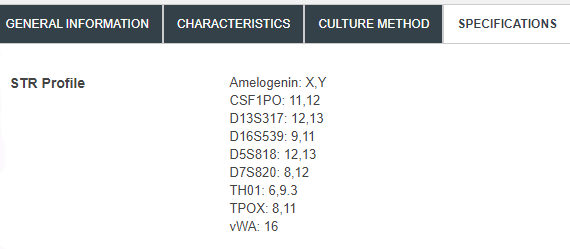
(图片来源:STR profile of FHC, ATCC)
● 目前ExPASy (Cellosaurus)、ATCC均已收录FHC的标准STR数据。ExPASy与ICLAC虽未将其标注为问题细胞,但鉴达的鉴定结果显示,FHC细胞株已出现被污染的情况。
1
FHC由K.M. Siddiqui、D.P. Chopra于1984年建株。细胞株来源于由阿拉巴马大学医院提供的合法流产胎儿(孕周大约13-17周)的结肠粘膜组织。
结肠与大肠、回肠等分离后,将其纵向剥离、用L15培养基进行数次冲洗以去除其他残留物。结肠粘膜从肌层轻轻刮下,转移至含0.1%胶原酶但无FBS的培养基中;粘膜切碎后在36.5℃、CO2与空气比为5%:95%的湿化气体条件下进行30min孵育;孵育后,对组织进行轻柔摇动、转移至含0.1%胶原酶的HBSSF培养基中、在磁力搅拌器上36.5℃条件下裂解5min;组织悬浮液600rpm下离心2min;上清液去除、沉淀用含0.1%胶原酶的HBSSF培养基重悬,并继续裂解15min;细胞悬浮液加入等量的含10%FBS培养基、80x g条件下离心7min;沉淀用含10%FBS培养基重悬;台盼蓝染色排除法计算细胞活力及密度。
将原代FHC细胞分别在35mm、60mm培养皿中进行接种。培养第一周,培养基24h换一次;此后48h换一次。另外,在培养过程中会出现星状成纤维细胞,需要经常用橡胶刮板将其去除。培养24h后,大部分贴壁细胞会出现独特形态特征:总体呈多边致密形态、细胞边缘光滑 、细胞间紧密粘附。PAS染色阳性结果显示细胞可合成粘多糖。电镜结果也显示细胞间的紧密粘附形态,主要通过桥粒、细胞表面的微绒毛建立胞间致密联系。原代培养细胞中也发现了肠道杯状细胞,除了具有典型的可分泌黏蛋白原的小囊泡,也具有和FHC细胞一样的桥粒和微绒毛。
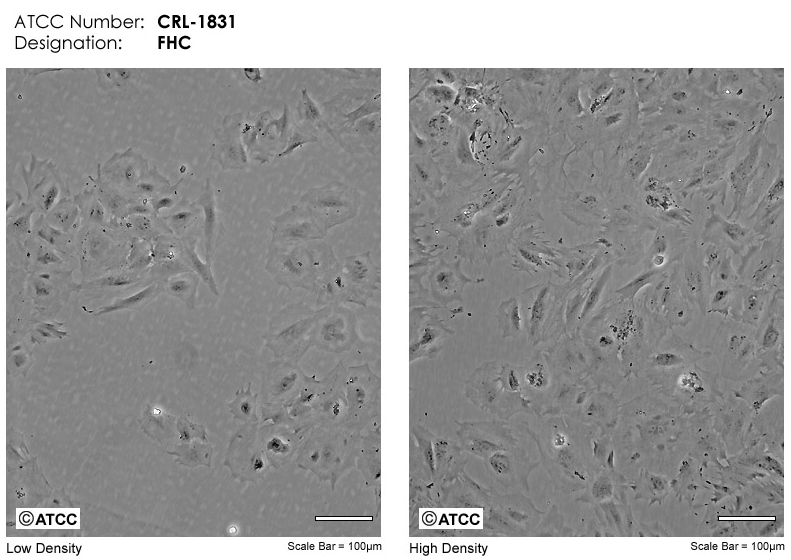
(图片来源:Images of FHC, ATCC)
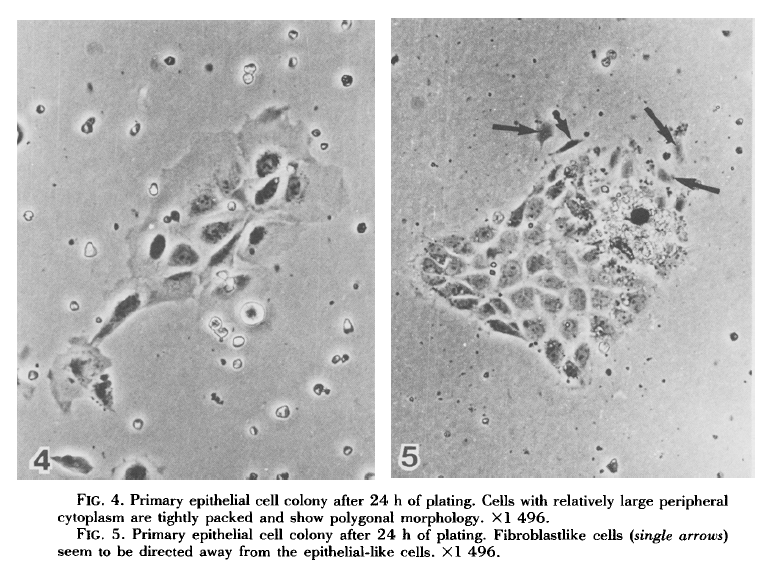
(图片来源:K.M.Siddiqui and D.P. Chopra, 1984)
原代细胞培养2-3周后,收获具有上皮细胞形态特征的细胞进行传代培养,一般接种后24h会再次贴壁。除了具有原代细胞的形态特征及PAS染色阳性结果,传代培养细胞还具有胞间指状突、致密染色质的锯齿状核、浓集趋边的异染色质。另外,溶酶体及多泡体在传代培养细胞可以观察到,但在原代细胞中则不常见到。
细胞生长动力学方面,通过对第13代细胞生长观测发现,FHC细胞株的倍增时间大约为62h。
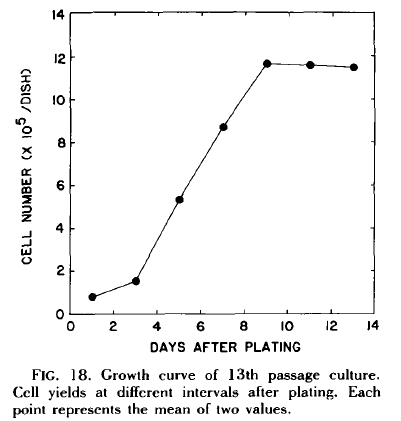
(图片来源:K.M.Siddiqui and D.P. Chopra, 1984)
2
自建株以来,FHC便常被用于各种基础实验中,主要集中于结肠癌发生机制、细胞分化调节及细胞凋亡等相关研究。2010年Karel Soucek 等人便发现,FHC细胞有相当数量的染色体异常、TP53基因也存在突变;另外细胞株在体外的半固体培养基上具有锚定非依赖性生长能力,在SCID小鼠体内能够形成固体瘤。这些独特的分子生物学方面的特性,让FHC成为更具价值的实验模型。
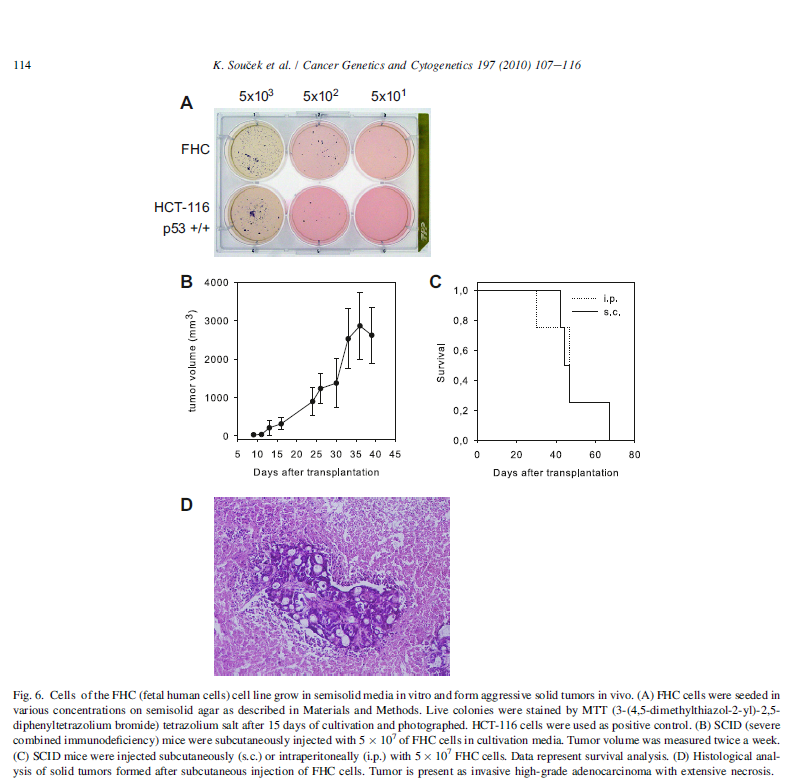
(图片来源:Karel Souceket al., 2010)
不过,随着在各实验室的培养传代、实验,以及实验室间的购买、交换,FHC细胞株逐渐被其他细胞污染。与其他被污染细胞株一样,2001年之前(STR分型还未逐步被应用到细胞株鉴定工作中)这种情况并未引起各方重视。目前FHC在Cellosaurus、ICLAC中未被标注、登记为问题细胞。但鉴达的FHC鉴定结果显示,FHC细胞株确实已经出现被污染的情况。
截止目前,鉴达受理FHC细胞株鉴定共计16株。与ExPASy、DSMZ等数据库重新比对确认后,实际鉴定结果显示:有10株为AGS,4株为HELA S3,2株为无匹配结果。鉴达所受理的16株FHC错误率达到了100%。
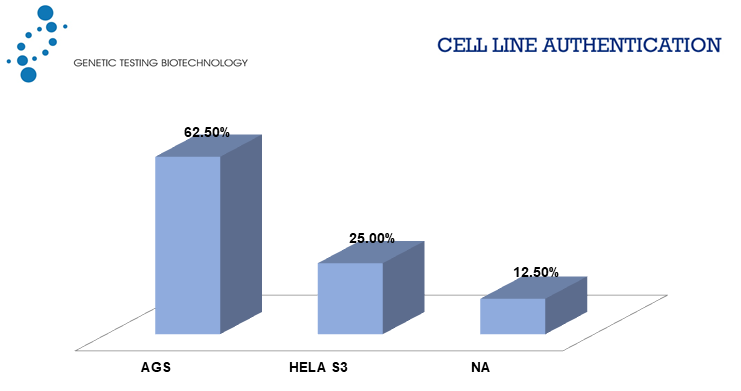
(图片来源:鉴达的FHC鉴定结果统计)
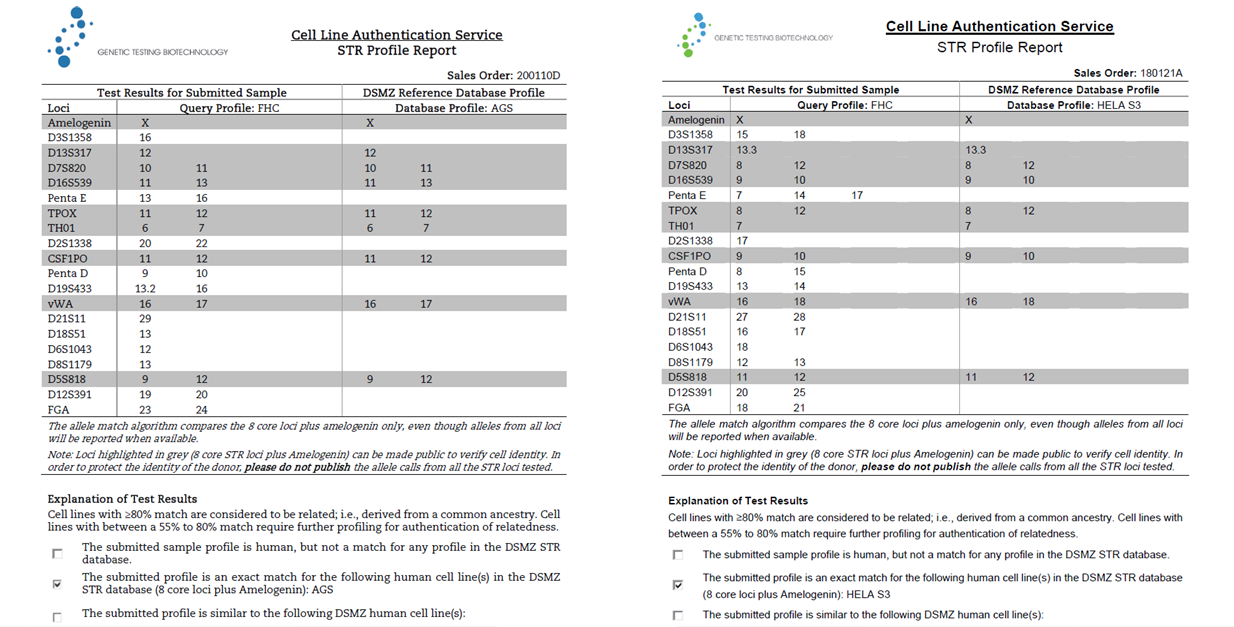
(图片来源:鉴达的FHC鉴定报告)
因此鉴达提醒各位老师,应谨慎使用FHC细胞株。若课题实验必须用FHC,鉴达建议大家,不管何种渠道的细胞株(实验室已有细胞或新采购买入的细胞)都应及时进行STR鉴定,以确认其身份。
参考文献:
1. Primary and long termepithelial cell cultures from human fetal normal colonic mucosa. InVitro. 1984 Nov;20(11):859-68. doi: 10.1007/BF02619632.
2. Fetalcolon cell line FHC exhibits tumorigenic phenotype, complex karyotype, and TP53 gene mutation. Cancer Genet Cytogenet. 2010 Mar;197(2):107-16.
苏州鉴达采用STR分型技术,针对人源细胞20个位点进行检测,3130XL型遗传分析仪进行毛细管电泳,与DSMZ、Cellosaurus、ATCC等数据库进行比对,出具权威英文报告,为全国近290家科研机构(高校、研究所、医院、公司)提供高质量服务,多家单位已利用鉴达细胞株鉴定服务在高水平期刊(如Clinical Cancer Research、Nature Commnications、Int J Cancer等)发表论文。
如需了解更多鉴达生物相关细胞株鉴定信息,可扫码关注官方公众号。将本文转发至朋友圈(至少保留三天)。添加微信或QQ客服后,将朋友圈截图发给微信或QQ客服,并备注单位及课题组(示例:XX大学XX课题组)。后期可有两次机会享七五折优惠,原价960元/株。
微信:18112556907、QQ:2791427499
标注优惠政策:
如客户在发表的高水平SCI论文(IF>8)中标注细胞株鉴定由我公司完成,我们承诺为客户免费检测两株细胞株。格式如下:The cell lines were characterized by Genetic Testing Biotechnology Corporation (Suzhou,China) using short tandem repeat (STR) markers.
Selected publications with Impact Factor >5.0 using our cell line authentication service
[1]. Ren A et al., Int J Cancer. 2019 Jul1;145(1):154-163.
[2]. Shen Y et al., Int J Cancer. 2019 Feb 1;144(3):651-664.
[3]. Xu H et al., Clin Cancer Res. 2019 Mar15;25(6):1923-1935.
[4]. Li B et al., Stem Cells. 2019 Feb 37(2):247-256.
[5]. Shi J et al., Mol Pharm. 2018 Sep4;15(9):4139-4147.
[6]. Li B et al., Oncogene. 2018 Oct 37(41):5508-5519.
[7]. Yuan Y et al., Int J Cancer. 2019 Apr1;144(7):1584-1595.
[8]. Yu J et al., J Hepatol. 2018 Jun 68(6):1214-1227.
[9]. Zhang N et al., Cell Death Differ. 2019 May 26(5):843-859.
[10]. Du L et al., Cell Death Dis. 2018 Jun18;9(7):714.
[11]. Zhou P et al., J Exp Clin Cancer Res. 2017Sep 2;36(1):115.
[12]. Wang X et al., Int J Cancer. 2017 Oct1;141(7):1422-1433.
[13]. Li Y et al., Nat Commun. 2017 Aug24;8(1):347.
[14]. Wang C et al., Carcinogenesis. 2017 Feb1;38(2):168-176.
[15]. Wang X et al., Cell Death Dis. 2016 Dec8;7(12):e2517.
[16]. Li P et al., Oncogenesis. 2017 Apr24;6(4):e322.
[17]. Du J et al., Mol Cancer Ther. 2018 Feb 17(2):419-431.
[18]. Chen L et al., Nat Commun. 2017 Jul17;8:16066.
[19]. Li H et al., Cancer Lett. 2018 Sep 28;432:121-131.
[20]. Zhang L H et al., Neuro Oncol. 2020 Jun 3;noaa138.
[21]. Ke Nie et al., Oncogenesis. 2020 May 15;9(5):51.
[22]. Li S J et al., Cancers (Basel). 2020;12(12):3864.
[23]. Zhang X L et al., Cancer Sci. 2020;111(10):3802-3812.
[24]. Hou X Y et al., Front Pharmacol. 2018;9:21.
[25]. Geng B et al., J Exp Clin Cancer Res. 2018;37(1):208.